- 日本語
- English
このページの使い方
EST データベース(マツノザイセンチュウ, ニセマツノザイセンチュウ, スギ, ヒノキ)
目次
データベースに格納されているデータ
ソースデータ
- EST(Expressed Sequence Tag)
- 本データベースの基礎となる配列データです。詳細は、各生物種の information を参照してください。
EST の配列には、次のような情報が関連づけられています。
- クローンID
- Genbank の Accession番号
- ライブラリの種類
- EST のシーケンス配列
- Cluster 番号と Subcluster 番号
- NCBI-nr
- NCBI 提供している全生物種の non-redundant な蛋白質配列です。
EST の Assemble 結果として得られる consensus 配列に、アノテーションを付与するため、すべてのコンセンサス配列をこの NCBI-nr にBLASTX した結果を格納しています。
コンセンサス配列に関連付けて、この BLASTX の結果の Top hit protein が データベースに格納されていますので、Top hit protein の description に文字列検索をかけて、目的の蛋白に類似性の高い遺伝子を見つけ出すことが可能となっています
データ処理方法
- EST(Expressed Sequence Tag)配列群を、 Partigene を利用して、Clustering & Assemble、及び、アノテーションを行います。
詳細は、Partigeneのサイトを参照してください。 -
- STEP1:塩基配列の相同性により、ESTを“Cluster(=遺伝子)”ごとに分類します。
- STEP2:各Clusterごとに、Phrap を利用して Assemble を行います。
- STEP3:Assemleの過程で、各 Cluster がさらに、小さなグループに分類らされますが、それを本データベースでは、SubCluster と呼んでいます。
その結果、Subcluster 単位に、次のデータを出力します。
Assemble で得られる Consensus 配列
Consensus 配列の素となる、EST配列とそのアライメント情報
- STEP4:すべての consensus 配列を NCBI-nr に BLASTX を行い、結果ファイルが出力されます。
- 上記の Clustering と Assemble の結果、NCBI-nr との BLASTX の結果、及び、各 EST の情報を postgreSQL のテーブルとして格納して、本データベースで検索できるようにしています。
データの構造と用語の説明
- 生物種のデータ構成
-
本データベースでは、生物種ごとに異なるデータ領域(PostgreSQLのデータベース)に格納しています。
従って、種間を越えた検索を行うことは原則としてできません。
Consensus配列へのBLAST検索は、可能です。
生物種は、アルファベット2文字を使います。
データベースは、<生物種表記><ビルド番号>で表します。
- Clusterの情報
-
Cluster;EST配列の相同性があるもの同士をグループ化したもの。
ClusterID:生物種のビルド単位に、ユニークな番号です。ビルド単位で異なる番号となります。表記は <生物種表記><ClusterID> - Subcluster の情報
-
Subcluster:ひとつの Cluster に属する EST を Assemble する過程で、一部配列の相同性がありますが、一部は相同性がない配列同士は、異なる Assemble が行われることがあります。
つまり、同じ Cluster に分類された配列がさらに小さなグループにわけられることになります。
このグループを本データベースでは、Subclusterと呼びます。
SuclusterID:PartiGene が付与した Cluster 内の通し番号です。
Consensus配列:Assemble の結果として得られた仮想最大長 cDNA 配列です。
Best hit protein:Consensus 配列を NCBI-nr(全生物種の既知蛋白質)に BLASTX を実行した結果のTop hit の蛋白質。
コンセンサス配列の機能予測結果です。
- EST配列の情報
-
CloneID:クローンの識別IDです。
AccessionNo.Genbank の Accession No.です。
mRNA source: クローンの由来情報です。
画面構成

画面説明
- [Cluster Search]
-
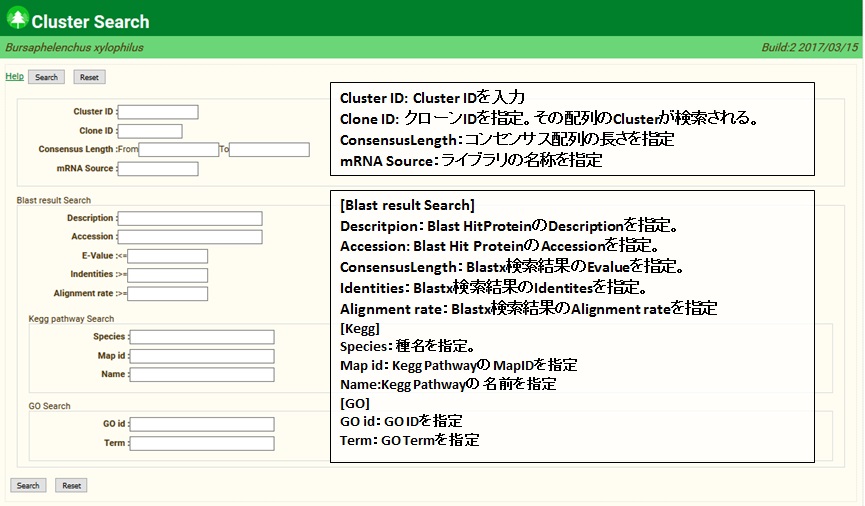
検索するための条件を入力して Seatch ボタンを押すと、検索条件に一致した Subcluster の一覧を表示します。
検索条件は、AND で結合されます。
- [Cluster List]
-
 結果一覧画面の ClusterID か Sucluster をクリックすると、Cluster Overview の Page が表示されます。
結果一覧画面の ClusterID か Sucluster をクリックすると、Cluster Overview の Page が表示されます。
Best Hit ProteinのEvalueをクリックすると、Protein Search Result のPageが表示されます。 - [Cluster Overview]
-

ひとつの Cluster の全構成要素を1pageに表示します。
この画面から Subcluster Viewer や、consensus 配列の NCBI-nr へのBLASTX結果画面を表示することができます。 - [Subcluster Viewer]
-
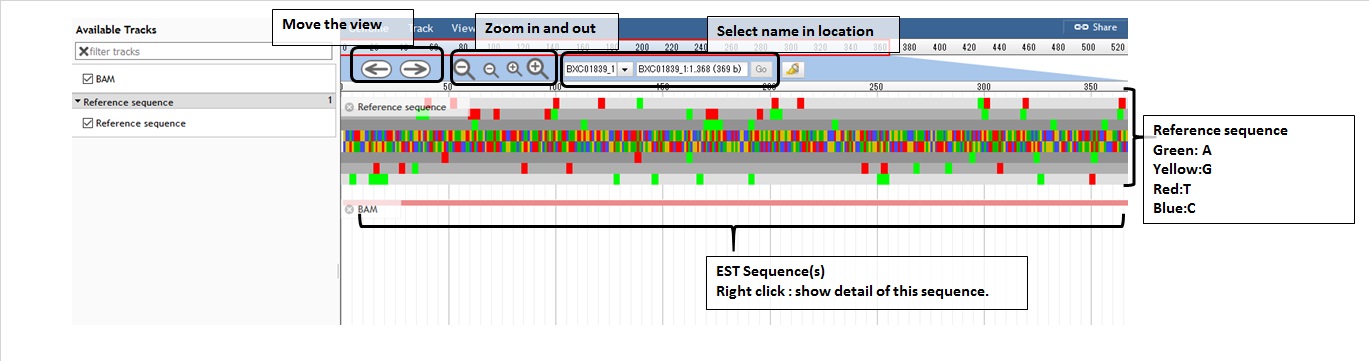 Subcluster のアライメントを表示します。
Subcluster のアライメントを表示します。
- [BLAST Search tool]
-

consensus 配列、及び、データソースとなった EST配列、NCBI-nr に BLAST検索を実行することができます。
また、他の生物種の配列に対しても、同時に BLAST検索ができます。 - [BLAST Search Result]
-
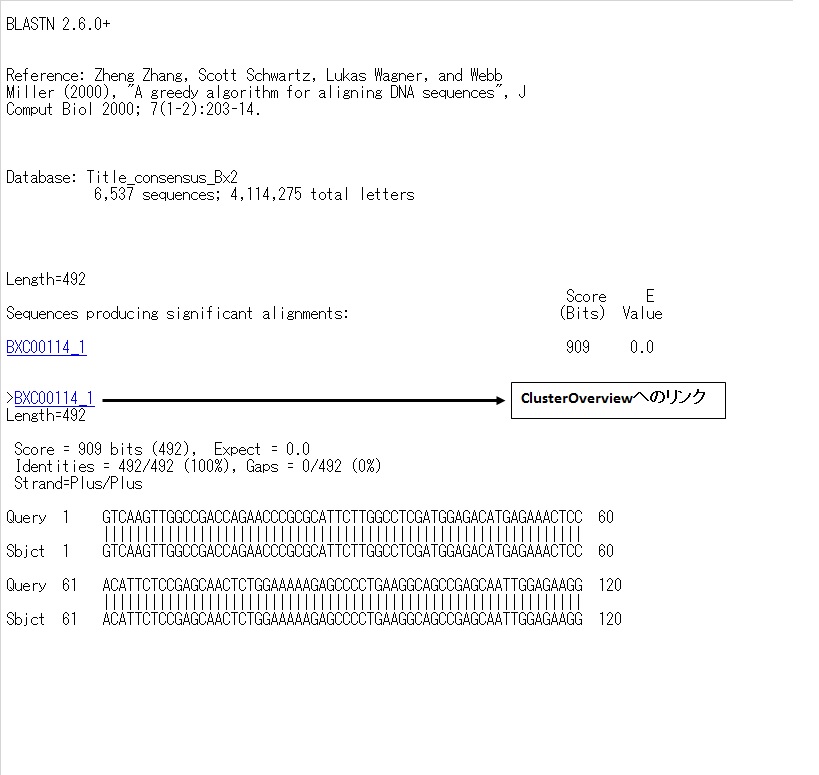
consensus 配列に BLAST を行った結果の画面で、consensus配列のIDをクリックするとCluster Overview画面に移行します。
GENOME データベース(シイタケ, マツタケ)
目次
データベースに格納されているデータ
ソースデータ
- ゲノムコンティグ
-
本データベースの基礎となる配列データです。上記ゲノムコンティグを、AUGASUTAS により遺伝子予測を行ないました。詳細は、各生物種の information を参照してください。
- NCBI-nr
-
NCBIが提供している全生物種の non-redundant な蛋白質配列です。
AUGASUTAS により得られた予測遺伝子配列を NCBI-nr に対して BLASTX を行い、
結果をアノテーションとして格納しています。
予測遺伝子配列に関連付けて、この BLASTX の結果の Top hit protein がデータベースに格納されていますので、Top hit protein の description に文字列検索をかけて、
目的の蛋白に類似性の高い遺伝子を見つけ出すことが可能となっています。
- NCBI gene データベース
-
NCGI が提供している XML ファイルを解析し、アノテーションとして使用しています。
- BLASTXの全ての結果
- KEGG Pathway
- Gene Ontology
- Pfam
-
AUGASUTAS により得られた予測遺伝子配列を NCBI-nr に対して BLASTX を行い、BLASTX 結果の全てとヒットしたGene Ontology、KEGG Pathway をデータベースに格納しています。
加えて、予測遺伝子配列と Pfam 検索の結果もデータベースに格納しています。
これらを条件に、予測遺伝子配列を検索することが可能となっております。 Pfam の詳細は、Pfam のサイトを参照してください。
データ処理方法
- 遺伝子予測
-
AUGASUTAS を使用しました。詳細はAUGUSTASのサイトを参照してください。
- 最適なトレーニングセットとコンティグの組み合わせを比較する為に次の3種類の生物種と2つのコンティグについて調査を行いました。
- トレーニングセット
- Laccaria: Laccaria bicolor(オオキツネタケ)
- Ustilago: Ustilago maydis(黒穂菌)
- Coprinus: Coprinus cinereus(ネナガノヒトヨタケ)
- コンティグ
- Le_contigs_s2.fasta 全33131コンティグ
- Le_contigs_oe_500_s2.fasta 500bp以上の12875コンティグ
- トレーニングセット
- 各トレーニングセット毎に予測された遺伝子数と遺伝子が予測されたコンティグの数
トレーニング
セット入力コンティグ 予測された
遺伝子数遺伝子が予測
されたコンティグ数Laccaria Le_contigs_s2.fasta 13089 7809 Le_contigs_oe_500_s2.fasta 12829 7544 Ustilago Le_contigs_s2.fasta 6566 4760 Le_contigs_oe_500_s2.fasta 6402 4591 Coprinus Le_contigs_s2.fasta 6366 4405 Le_contigs_oe_500_s2.fasta 6252 4286 - 採用したトレーニングセットとコンティグ
- トレーニングセット
- Laccaria: Laccaria bicolor(オオキツネタケ)
- コンティグ
- Le_contigs_s2.fasta 全33131コンティグ
- トレーニングセット
- 最適なトレーニングセットとコンティグの組み合わせを比較する為に次の3種類の生物種と2つのコンティグについて調査を行いました。
- NCBI の KEGG, GO 情報の XML を解析
-
- 入手元 URL
- ftp://ftp.ncbi.nih.gov/gene/DATA/ASN_BINARY/All_Data.ags.gz
- 入手日
- 2016年12月
-
-
XMLファイルをスクリプトにより解析し、データベース用のデータ化を行なう。
- 入手元 URL
画面構成
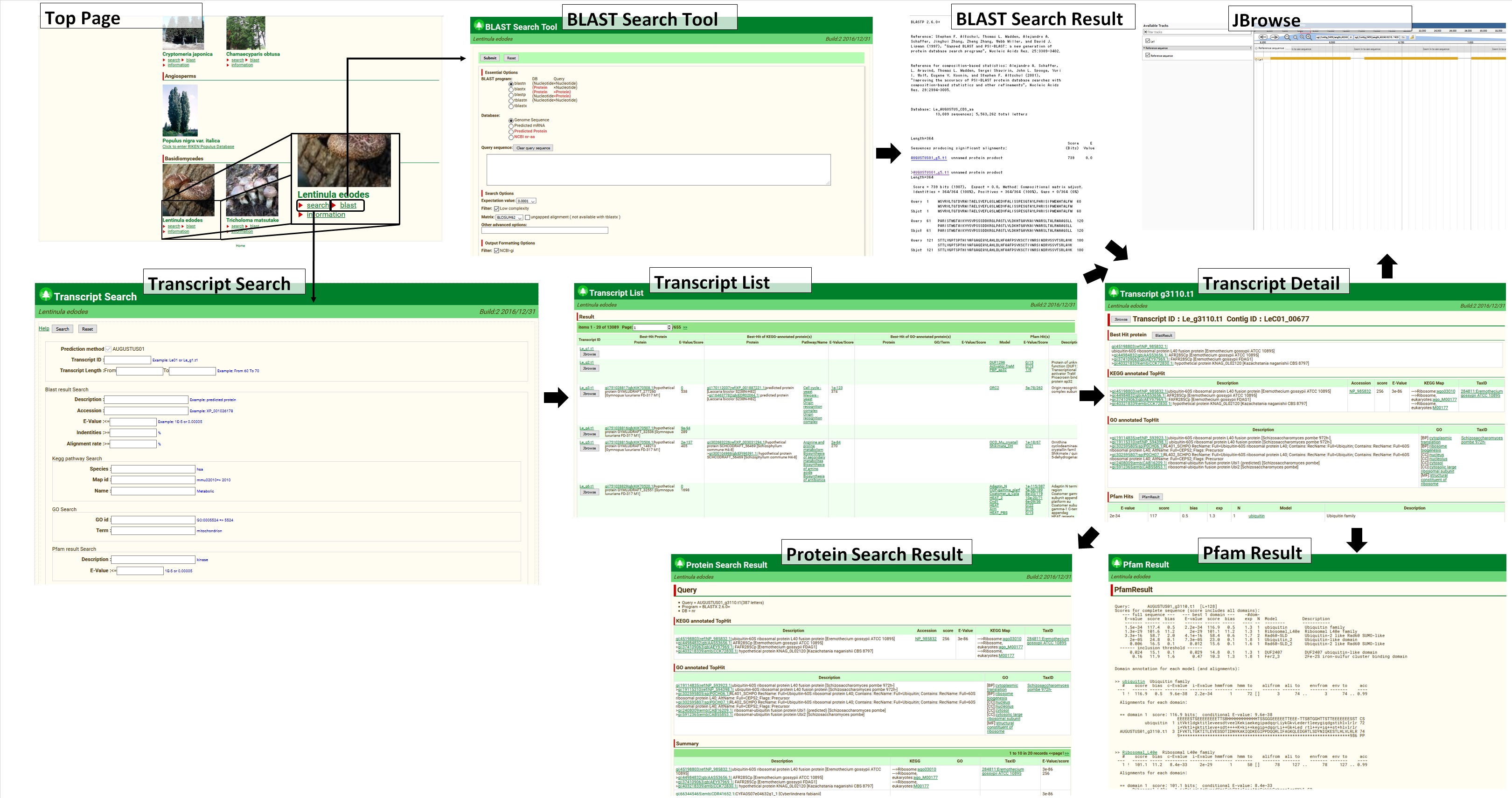
画面説明
- [Transcript Search]
-
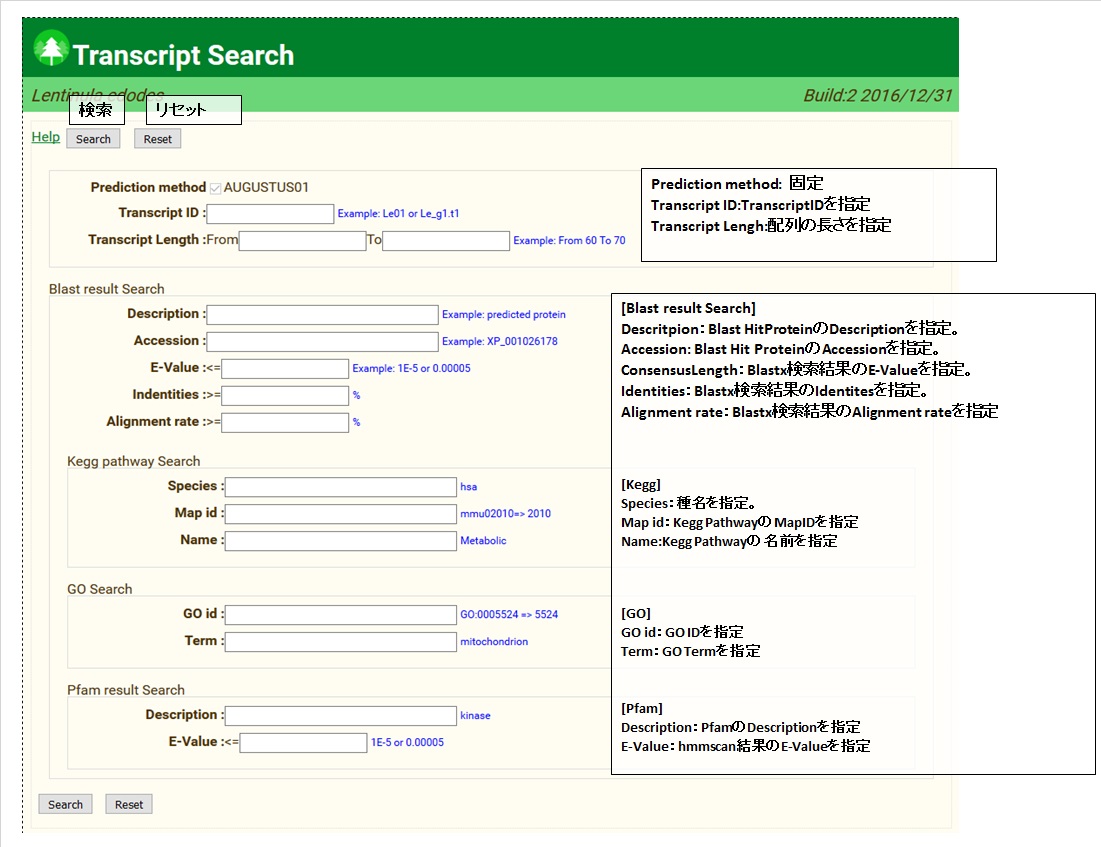
検索するための条件を入力して Seatch ボタンを押すと、検索条件に一致した予測遺伝子の一覧を表示します。(何も入力せず全件表示も可能)
検索条件は、AND で結合されます。
Help をクリックすると、Help の Page が表示されます。
- [Transcript List]
-

予測遺伝子の一覧を表示します。
Transcript ID をクリックする事により Transcript Dretail 画面を表示する事が可能です。
JBrowse ボタンを押すと JBrowse 画面を表示可能です。
Best-Hit ProteinのE-Value をクリックすると Protein Search Result 画面が表示されます。
Pfam HitsのE-Value をクリックすると Pfam Result 画面が表示されます。
・Transcript ID の命名規則
Le_g1.t1
Le: Lentinula edodes 生物種名
g1:gene00001 transcript予測遺伝子番号
.t1:transcript00001 transcript番号
- [Transcript Detail]
-

予測遺伝子について詳細な情報を表示します。
JBrowse ボタンを押すと JBrowse 画面を表示可能です。
Blast Resultボタンを押すと Protein Search Result 画面が表示されます。
Pfam Resutlボタンを押すと、Pfam Resultが表示されます。
- [Protein Search Result]
-

AUGASUTAS により得られた予測遺伝子配列を NCBI-nr に対して BLASTX を行なった結果が参照できます。
ページを下部までスクロールすると、Hit情報が順に表示されます。
- [Pfam Result]
-

AUGASUTAS により得られた予測遺伝子配列を Pfamに対して hmmscan を行なった結果が参照できます。
- [JBrowse]
-
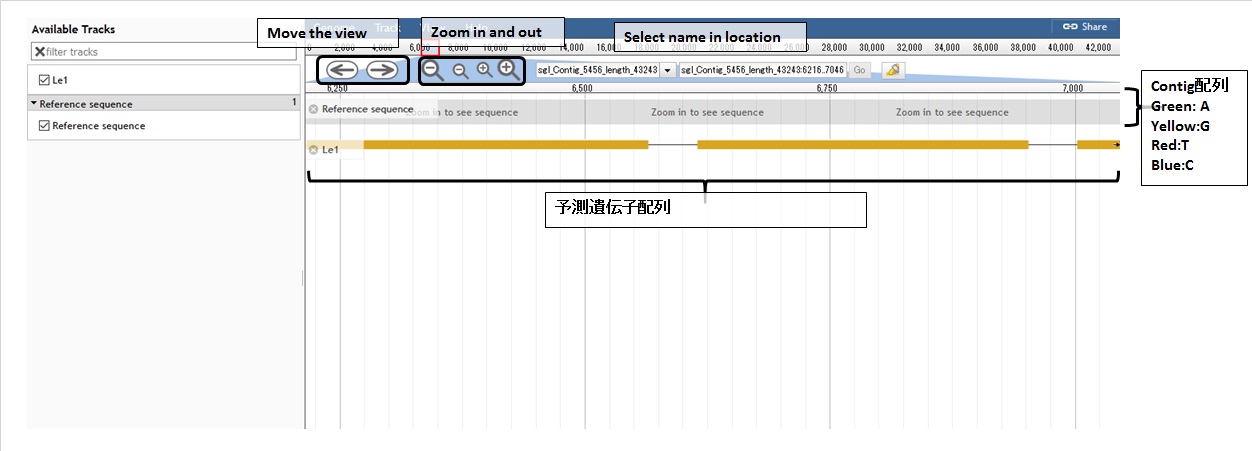
Contig配列と予測された遺伝子配列の情報が表示されます。
右クリックで、詳細が表示されます。
- [BLAST Search Tool]
-
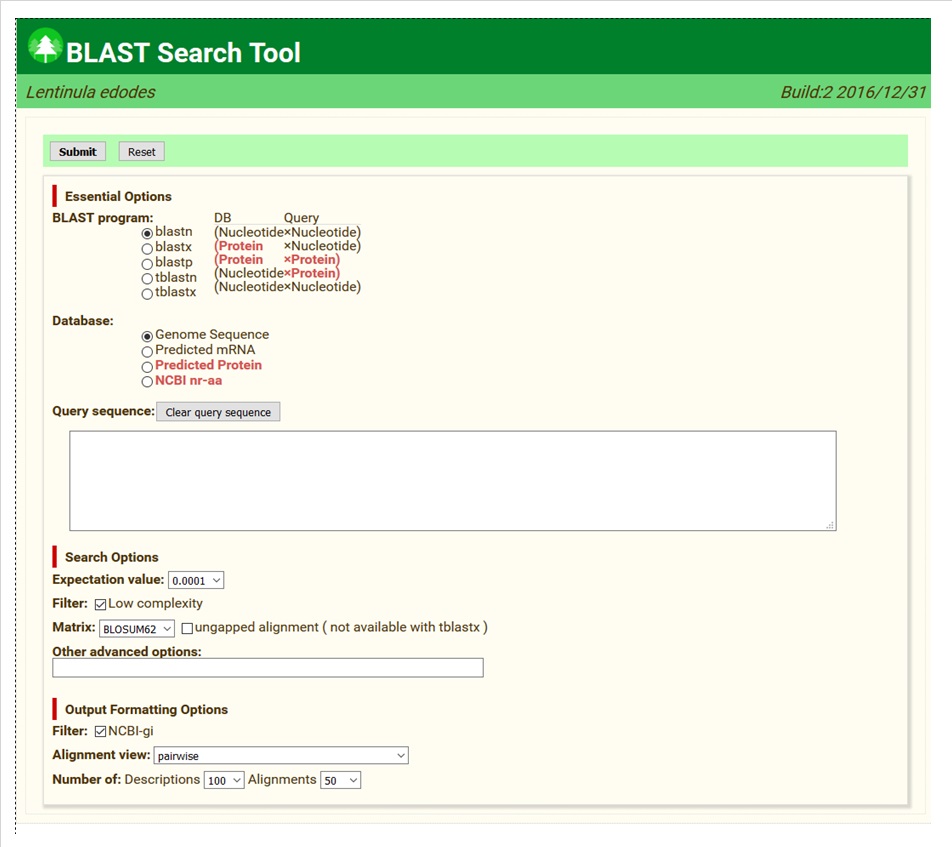
Query sequence に問合せ配列を入力し、Blast プログラムの選択、対象データベースの選択を行いBlast 実行ボタンを押すと Blast が行なわれます。
- [BLAST Search Result]
-

BLAST 結果画面で各 Alignment 情報のリンクをクリックするとTranscript Dtail 画面、またはNCBI-nr の場合、NCBI Protein Database に遷移します。
 ForestGEN
ForestGEN